FAQs¶
Q1. 如何删除分子中的Atom Map ID 信息?¶
A1. 借助于atom的ClearProp方法可以删除指定定属性的信息
示例代码:
from rdkit import Chem
smi='[CH3:1][C:2]([N:4]([C:9](=[O:10])[CH3:8])[CH2:5][CH2:6][NH2:7])=[O:3]'
m=Chem.MolFromSmiles(smi)
for a in m.GetAtoms():
a.ClearProp('molAtomMapNumber')
# [a.ClearProp('molAtomMapNumber') for a in m.GetAtoms()]
newsmi = Chem.MolToSmiles(m)
print(newsmi)
输出:
CC(=O)N(CCN)C(C)=O
Q2. 如何显示分子的kekulize的smiles形式?¶
RDKit 默认会计算分子的芳香性,因此要想输出分子的kekulize的smiles,需要删除分子中的芳香性信息。 然后再输出smiles。
示例如下:
from rdkit import Chem
mol = Chem.MolFromSmiles('C=Cc1ccccc1')
smi1 = Chem.MolToSmiles(mol,kekuleSmiles=True)
Chem.Kekulize(mol, clearAromaticFlags=True)
smi2 = Chem.MolToSmiles(mol,kekuleSmiles=True)
print("smi1",smi1)
print("smi2",smi2)
输出:
smi1 C=CC1:C:C:C:C:C:1
smi2 C=CC1=CC=CC=C1
Q3. 如何批量计算ECFP4指纹的01字符串形式?¶
参考脚本如下:
from rdkit import Chem
from rdkit.Chem import AllChem
from tqdm import tqdm
fh = open('smiles.csv')
fw = open('smiles_ecfp4.csv','w')
for line in tqdm(fh.readlines()[0:]):
name,smi=line.strip().split(',')
#print(smi)
m1 = Chem.MolFromSmiles(smi)
#print(m1)
if m1:
ecfp4 = AllChem.GetMorganFingerprintAsBitVect(m1, radius=2, nBits=2048)
ecfp4bit= ecfp4.ToBitString()
else:
ecfp4bit='None'
newline="%s,%s,%s\n"%(name,smi,ecfp4bit)
fw.write(newline)
fw.close()
Q4. 如何根据化合物的原子ID提取化合物的片段?¶
示例分子如下:
from rdkit import Chem
smi = 'O=C(OC)C1=CC=CC=C1'
mol = Chem.MolFromSmiles(smi)
for atom in mol.GetAtoms():
atom.SetProp('atomLabel',str(atom.GetIdx()))
mol
输出:
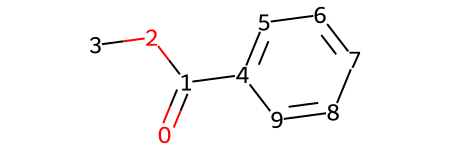
我们希望提取苯环的片段,原子ID是4-9.
代码如下:
from rdkit.Chem import AllChem
fragsmi= AllChem.MolFragmentToSmiles(mol,[4,5,6,7,8,9])
fragmol=Chem.MolFromSmiles(fragsmi)
fragmol
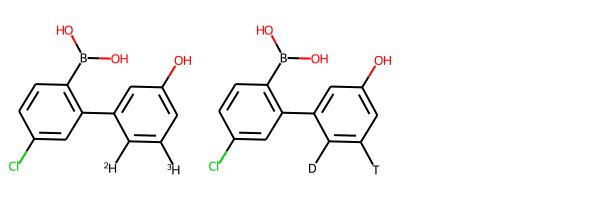
Q5. 如何显示H的同位素为DT?¶
示例代码:
import rdkit
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem.Draw import MolsToGridImage
smi='[3H]C1=C([2H])C(C2=C(B(O)O)C=CC(Cl)=C2)=CC(O)=C1'
mol=Chem.MolFromSmiles(smi)
mol1=Chem.MolFromSmiles(smi)
for atom in mol.GetAtoms():
if(atom.GetIsotope()==3):
atom.SetProp('atomLabel','T')
if(atom.GetIsotope()==2):
atom.SetProp('atomLabel','D')
MolsToGridImage([mol1,mol])
输出: