常用小技巧888¶
- 如何在Jupyter NoteBook 中直接显示分子对象为结构图片
from rdkit.Chem.Draw import IPythonConsole #Needed to show molecules
mol
from IPython.display import display
for mol in mols:
display(mol)
- RDKit如何给复杂分子绘制漂亮的2D结构?
建议更新RDKit版本,同时指定开启新的2D坐标产生的算法。 默认采用的是schrodinger中的2D 算法。
from rdkit.Chem import rdDepictor
rdDepictor.SetPreferCoordGen(True)
Chem.MolFromSmiles('CC1CCC2C(C(=O)OC3C24C1CCC(O3)(OO4)C)C')
参考: https://github.com/rdkit/rdkit/issues/2960
- 如何按照表格方式绘制多个分子?
绘制多个分子的时候推荐 Draw.MolsToGridImage 方法, 图片清晰,布局美观。
from rdkit import Chem
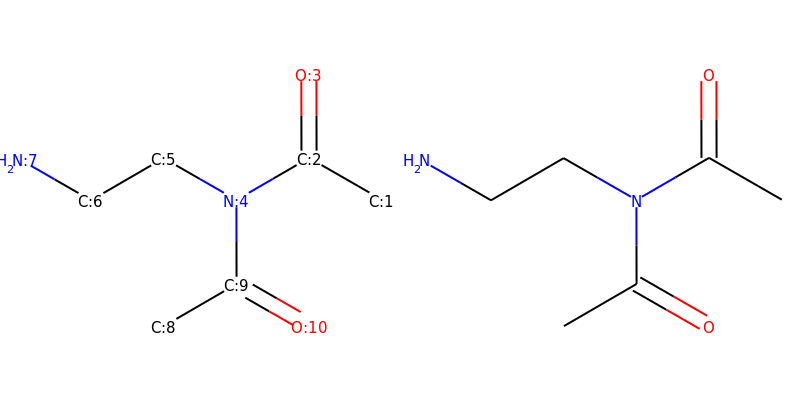
smi='[CH3:1][C:2]([N:4]([C:9](=[O:10])[CH3:8])[CH2:5][CH2:6][NH2:7])=[O:3]'
mol=Chem.MolFromSmiles(smi)
# display(mol)
for a in mol.GetAtoms():
a.ClearProp('molAtomMapNumber')
newsmi = Chem.MolToSmiles(mol)
mol2=Chem.MolFromSmiles(newsmi)
mol1=Chem.MolFromSmiles(smi)
mols = [mol1,mol2]
Draw.MolsToGridImage(mols,molsPerRow=2,subImgSize=(400,400),legends=['' for x in mols])
输出:
- 如何删除分子中atomMapId 信息?
示例代码
smi='[CH3:1][C:2]([N:4]([C:9](=[O:10])[CH3:8])[CH2:5][CH2:6][NH2:7])=[O:3]'
mol=Chem.MolFromSmiles(smi)
[a.ClearProp('molAtomMapNumber') for a in mol.GetAtoms()]
Chem.MolToSmiles(mol)
输出:
'CC(=O)N(CCN)C(C)=O'
- 如何关闭RDKIT warnings警告?
from rdkit import RDLogger
RDLogger.DisableLog('rdApp.*')
